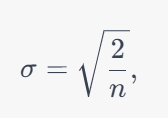728x90
반응형
1. Intro
- 본 논문에서는 CNN의 성공이 Training Set의 양이 커지면서 생긴 제한적인 이유라고 말함.
- 이전까지는 CNN은 Classification을 위해 많이 사용되었으나 생물학 분야의 영상 처리에서는 Localization이 중요했고, Semantic Segmentation의 중요도가 높았음.
- 하지만 생물학에 대한 Sample의 개수가 1000개밖에 되지 않는 것이 다수.
- 기존에 사용하던 sliding-window 2가지 단점

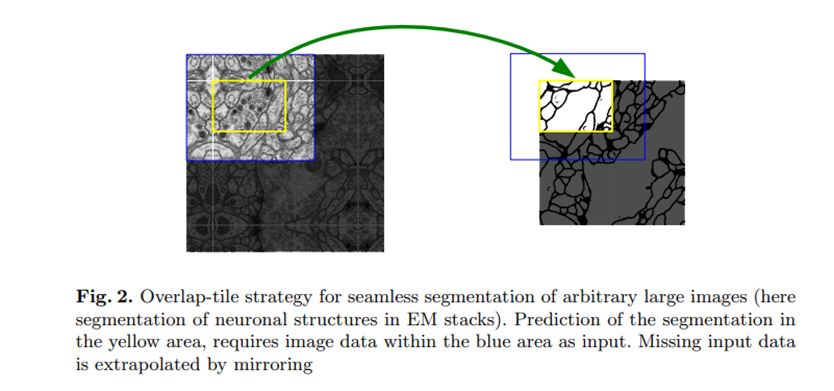
- redundancy of over lapping patch(겹치는 패치의 불필요한 중복성)위의 사진에서 보이는 것과 같이 patch를 옮기면서 중복이 발생하게 됨=> 이 중복된 부분은 이미 학습된(검증된) 부분을 다시 학습하는 것이므로 똑같은 일을 반복하는 것과 같음. 즉, 불필요한 중복에 대한 내용도 학습하기 때문에 속도도 느리고 시간도 오래 걸림
- trade-off between localization accuracy and use of context patch 사이즈가 크면, max pooling이 더 많이 적용 되고 정확한 위치 정보를 알기에는 어렵지만, 더 넓은 범위의 이미지를 보기 때문에 context 인식에는 효과를 가짐.
⇒ 이를 해결하기 위해 본 논문에서는 Fully Convolutional network를 소개함.
1.5 U-net: Improved Sliding Window Search Method - input
1.5 U-net: Overlap tile Method (Strategy) - input
2. Network Architecture

- 전체 네트워크: 23 conv
- Contraction Path
- CNN 이미지의 context를 포착할 수 있도록 해줌.
- Expansive path
- 작아진 feature map을 Upsampling 해서 원본 이미지와 비슷한 크기로 늘려준 후, Contracting Path의 feature map과 결합하여(회색 화살표 부분) 더 정확한 위치 정보를 가진 segmentation map을 얻게 됨.
- Contraction path

- 파란색 부분: conv
- 두번의 3x3 conv
- ReLU 사용
- 빨간색 부분: max pooling
- 2x2 max pooling 사용
- channel 2배씩 늘려줌
- Expansive Path

- 초록색 부분: up-sampling
- 크기를 키워줌 (2배씩)
- 파란색 부분: conv
- 두번의 3x3 conv
- ReLU 사용
- 회색 부분: concat
- contraction path에서 추출된 feature map (경계 부분을 crop) 을 concat
- 좌우반전 → 확장

- conv 연산 수행할 때, 입력 이미지의 경계 부분은 커널이 겹쳐지지 않기에 출력 이미지에서 손실될 수 있다는 것을 의미
- 즉, 보통 3x3, 5x5 7x7 필터를 사용하는데, 가장자리 부분은 겹치는 부분이 거의 없어서 정보가 손실된다고 표현함
- 청록색 부분: 1x1 conv
- 마지막은 class를 2로 설정 (배경vs세포)
- 여기서 x와 y는 입력 이미지의 가로와 세로 크기를 의미함.
- 최대 풀링 연산은 일반적으로 입력 이미지 크기의 절반으로 줄어드는데, 이 때 입력 이미지의 크기가 홀수일 경우 정확히 반으로 나누기가 어려워짐.
- 따라서 입력 이미지의 가로와 세로 크기가 짝수인 경우, 최대 풀링 연산이 반으로 줄어들었을 때도 모든 레이어에서 적용되도록 할 수 있도록 함.
- 파란색 부분: conv

3. Training

- 현미경으로 찍은 사
- color을 다르게 한 (정답 답지- ground truth)
- segmentation을 black and white로 만든 결과값
- 세포 경계선 학습시킨 이미지
- 경사하강법 사용
- GPU 메모리를 최대한 활용하기 위해 배치 사이즈를 줄이고, 패치를 크게 했음
- 하지만, 배치 사이즈가 작은 경우, 최적화 잘 안됨
- 이를 보완하기 위해 모멘텀을 0.99로 → 과거의 값이 많이 반영되도록.
- softmax

- 전체 클래스 중 해당 클래스일 확률값

- 에너지 함수: 최종 피쳐 맵에 대한 픽셀 단위의 소프트맥스와 교차 엔트로피 손실 함수의 결합으로 계산됨.

- 세포 사이에 간격이 짧아서 세포별로 구별이 힘든 경우가 있기 때문에, 거리가 가까울수록 가중치를 크게 해서 분리를 확실하게 해버림.
- 세포 사이에 떨어진 간격이 짧아 세포별로 구별이 힘든 경우가 발생하기 때문.
- wc: 각각 클래스마다 등장하는 빈도수 조율( ex: 배경과 세포의 등장 횟수 )
- d1(x): 첫번째로 가장 가까운 세포까지의 거리 → 여기서 x는 두 세포 사이에 존재하는 좌표값
- d1(x): 두 번째로 가까운 세포까지의 거리
- σ=5, w_0 =10
++ 거리가 짧을수록 가중치를 크게
ex) d1=2, d2=4인 경우, 값은 0.00000152299
d1=1, d2=3인 경우, 값은 0.00335로 거리가 짧을수록 가중치가 크게 됨
즉, 이 말은 세포 간의 분리를 확실하게 하겠다!의 의미를 가질 수 있음 (분리를 더 잘하도록)
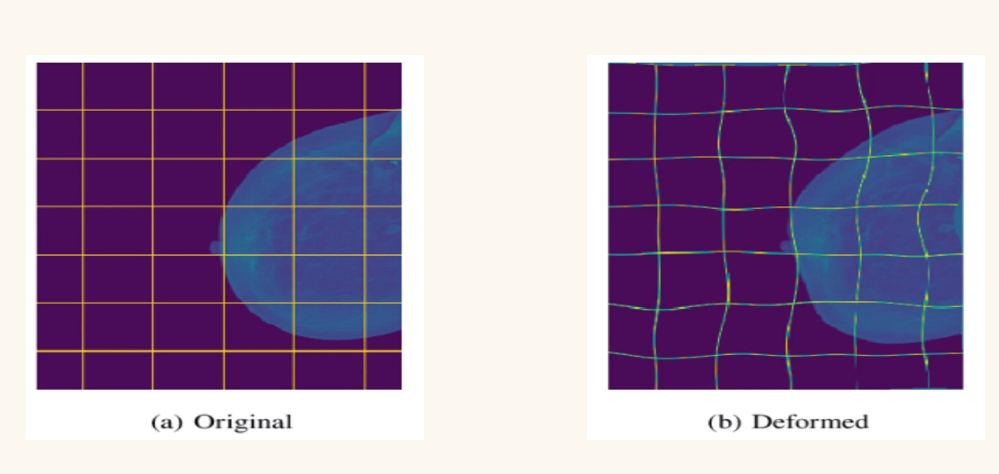
3.1 (Training) Data augmentation
4. Experiments
4.1 EM segmentation challenge
- 우선 U-Net은 전자현미경으로 관찰되는 뉴런 구조에서 cell segmentation task를 수행.
- EM segmentation challenge에서 제공되는 데이터셋으로 학습.
- 데이터셋은 전자 현미경으로 찍은 512 x 512 해상도의 이미지 30장으로 이루어져 있고 이미지의 각 부분에 세포는 흰색으로, 세포막(membrane)은 검은색으로 색칠한 ground truth segmentation map을 만듬.
- 테스트용 이미지도 있는데 ground truth segmentation map은 공개되지 않았다고.
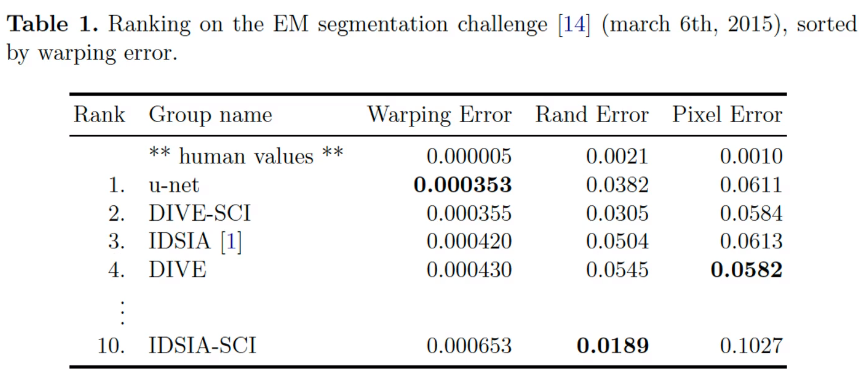
- U-net이 가장 좋았다!
4.2 ISBI cell tracking challenge

- a, c가 입력 이미지고 b, d가 ground truth segmentation map
- a, c와 같이 광학 현미경에서 얻은 이미지로 b, d와 같이 세포를 구별하는 task를 U-Net이 얼마나 잘 수행하는지 시험해봄.

- U-net이 좋았다!
5. Conclusion
- U-Net은 다양한 biomedical segmentation applications에서 "아주" 좋은 성능을 보여줌
- 저자는 elastic deformation이 포함된 Data augmentation 덕분에 적은 사이즈의 데이터셋만 요구했고 합리적인 학습 시간(NVidia Titan GPU (6 GB)에서 10시간 학습)을 가졌다고 말함.
- 그리고 마지막으로 U-Net의 구조가 다양한 task에 쉽게 응용될 수 있을거라 확신한다고 말하며 논문을 끝마침.
6. Reference
728x90
반응형
'Deep Learning > [논문] Paper Review' 카테고리의 다른 글
| Inception-v4, Inception-ResNetand the Impact of Residual Connections on Learning (0) | 2023.07.05 |
|---|---|
| Seq2Seq (0) | 2023.07.05 |
| Bert (0) | 2023.07.05 |
| VIT [AN IMAGE IS WORTH 16X16 WORDS: TRANSFORMERS FOR IMAGE RECOGNITION AT SCALE] (0) | 2023.07.05 |
| RetinaNet (0) | 2023.07.05 |